单细胞转录组测序产生的数据是成百上千个基因在上万个细胞中的表达情况,属于高维数据,我们需要对数据进行严格的质控与过滤,将合格的数据降维到低维子空间,使数据可视化。
上一期已经带大家了解了scRNA-Seq数据的预处理,那么本期,小编就来介绍一下数据分析的标准化与聚类分析。
一、上海烈冰科技数据分析流程介绍
二、工具介绍
Seurat_Normalized(标准化)——采用Seurat package对数据进行过滤并校正批次效应(Batch Effect),采用PCA算法及tSNE算法对基因表达矩阵进行降维处理和信息可视化展示。
Seurat_Cluster(聚类分析)——根据基因表达的情况,通过无监督聚类算法(Graph-based clustering或k-means clustering) 将降维后的细胞聚类分群。再通过Wilcoxon秩和检验分析计算出不同细胞类群的标识基因(Marker Gene),并对所属细胞类群进行推测和鉴定。
三、结果展示
(一) 数据标准化
1.线粒体RNA占比:
由于Dead Cell胞内RNA会流出,线粒体RNA占比会随之增高,因此我们一般通过设定线粒体RNA占比阈值来过滤Dead Cell。

左图描述了每个细胞线粒体RNA占比—UMI数量相关性,红色和黑色圆点代表两个样本的细胞。X轴代表每个细胞对应的UMI数量,Y轴代表每个细胞线粒体RNA占比;右图为细胞线粒体RNA占比分布的Violin图。
图中线粒体RNA占比阈值建议设为0.2,线粒体RNA占比超过0.2的细胞认为是Dead Cell,可将其过滤掉。当然,不同类型的细胞线粒体RNA占比也不同,例如心肌细胞、肝细胞等高代谢、凋亡类细胞,其线粒体RNA占比相对较高。因此,需要结合具体的细胞类型来最终决定线粒体RNA占比的阈值。
2.细胞的基因数量:
我们一般通过设定细胞的最小基因数量去除假细胞和低质量细胞(Low Quality Cell);通过设定细胞的最大基因数量可以一定程度上去除双细胞(Doublet Cell)。
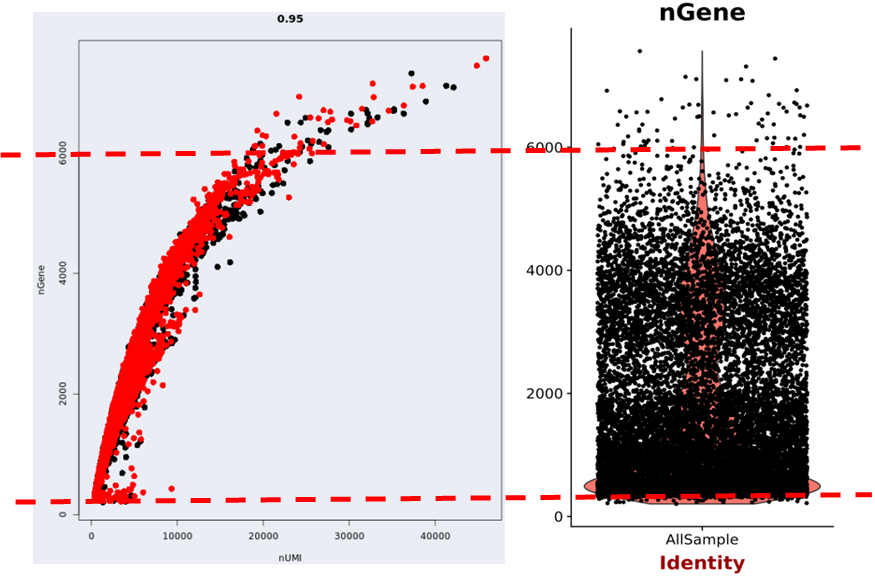
左图为每个细胞的基因—UMI数量相关性分析图,右图为每个细胞基因数量分布的Violin图。图中基因数量阈值建议设定为200-6000,可有效去除假细胞、低质量细胞和双细胞。
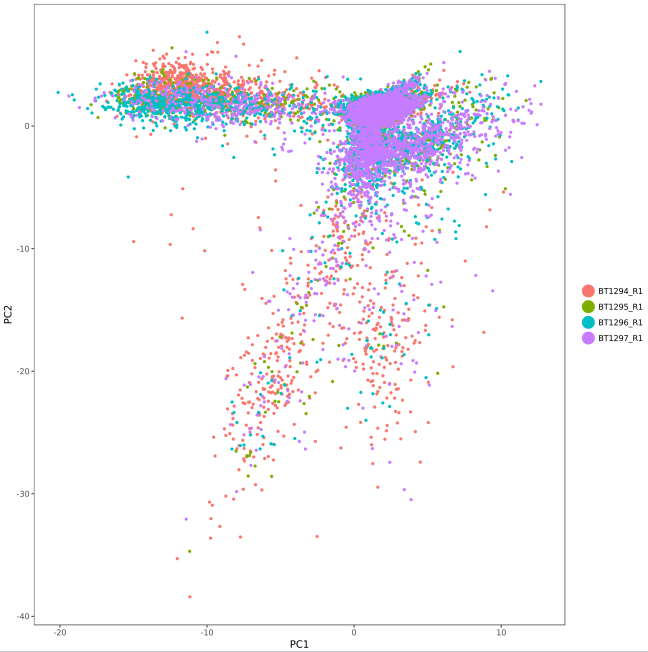
3.PCA分析:
该图主要描述了不同样本中所有细胞在PC1和PC2(即主成分分析中影响最大的两个主成分)所组成的面中的定位情况。
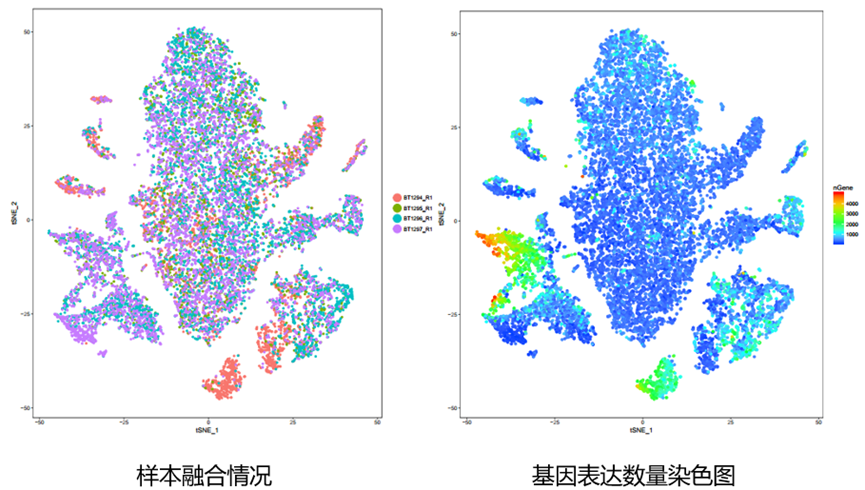
4.t-SNE图:
主要展示了每个样本中所有细胞的tSNE定位情况、样本融合情况,以及通过基因数量、UMI数量、线粒体RNA占比进行染色的情况,如下所示:
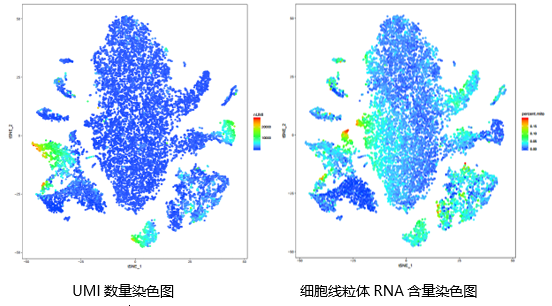
(二)聚类分析
1.细胞分群的t-SNE图:
该图代表t-SNE定位并基于Graphcluster或者KMean算法无监督聚类后的分群情况。
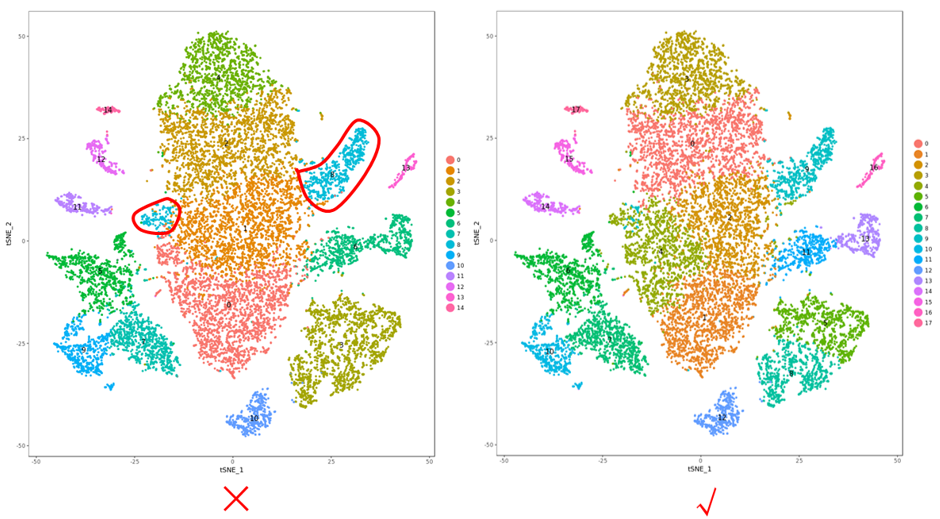
左图中cluster8被其他群分割成两部分,这样的t-SNE分群结果并不是特别理想。建议调整分析时的resolution参数,将其调大,使分群更加细致。将resolution参数由0.8调至1.0,得到右图较好的t-SNE分群结果。
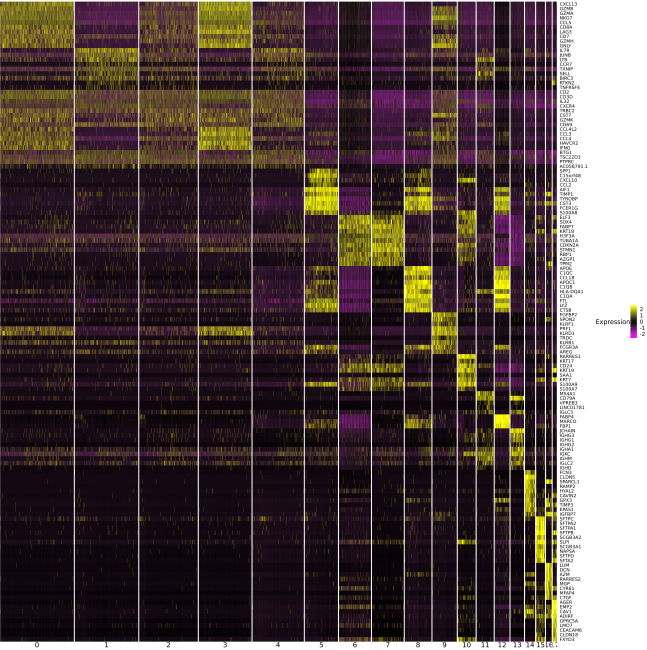
2. Top20 Marker基因的Heatmap:
可观察特定marker基因在不同cluster的表达差异,以此初步判断细胞类型及类群合并。采用细线区分不同的Cluster,颜色深浅代表基因表达高低,其中黄色为高表达,暗红色为低表达。根据各cluster的基因表达的整体类似度,下图可以初步判断将cluster0、3合为一群,cluster6、7合为一群,cluster5、8、12合为一群。
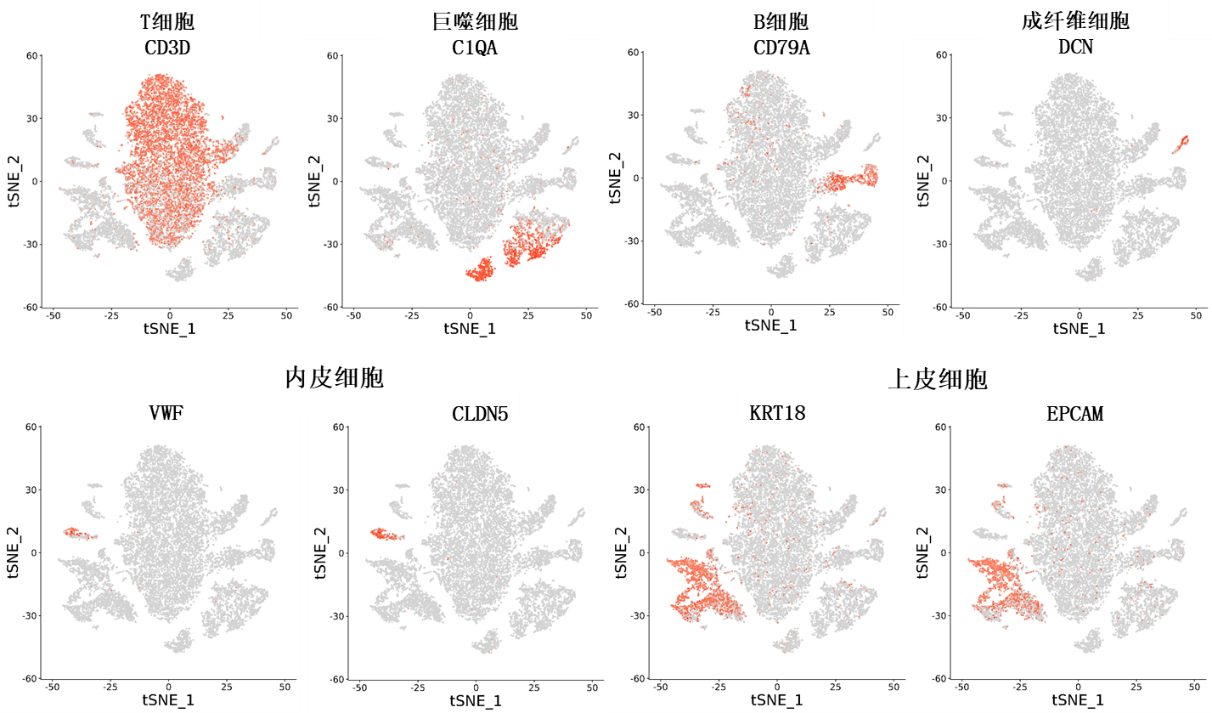
3.Feature plot:
该图主要描绘了选定Cluster中 Marker 基因在所有单细胞中的分布情况,更加直观的了解marker基因的整体表达情况,用于判定cluster所属的细胞类型。根据经典特异性marker基因的Feature plot,可以初步判断出T细胞(CD3D)、巨噬细胞(组织样本C1QA)、B细胞(CD79A)、成纤维细胞(DCN)、内皮细胞(VWF、CLDN5)及上皮细胞(KRT18、EPCAM)。
4.细胞类型鉴定结果tSNE图:
下图就是原始的tSNE分类结果,共有18个类群,此时要结合第3步中Marker基因特异性表达的结果对Cluster进行合并。
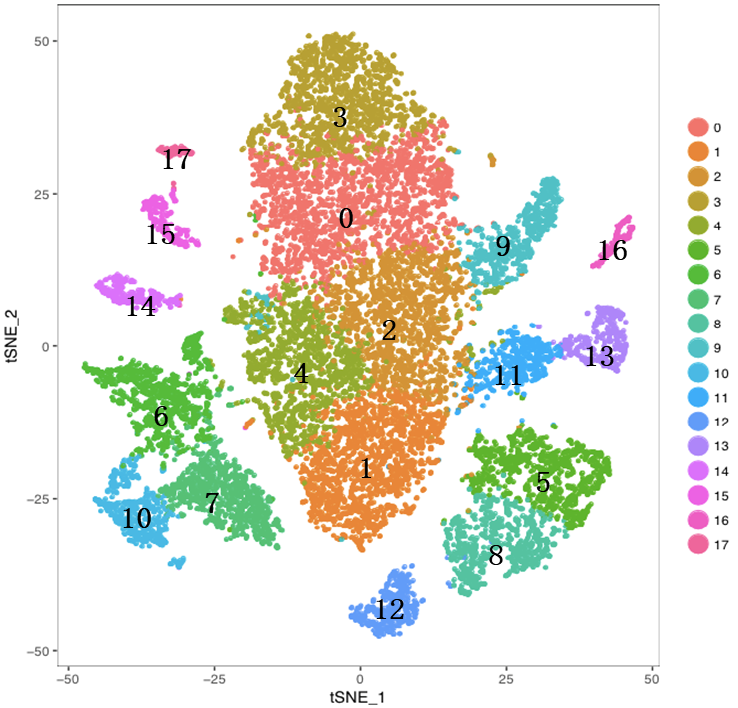
5.细胞亚型分群图:
该图将0、1、2、3、4、9 Cluster合并为T细胞(CD3D);将5、8、12 Cluster合并为巨噬细胞(组织样本C1QA);将11、13 Cluster合并为B细胞(CD79A);16 Cluster为成纤维细胞(DCN);14 Cluster为内皮细胞(VWF、CLDN5);将6、7、10、15、17 Cluster合并为上皮细胞(KRT18、EPCAM)。
后续可以将自己关注的细胞亚型再次细分,并进行其功能性分析,为解读生物学意义细节提供基础,后续讲解会详细介绍。
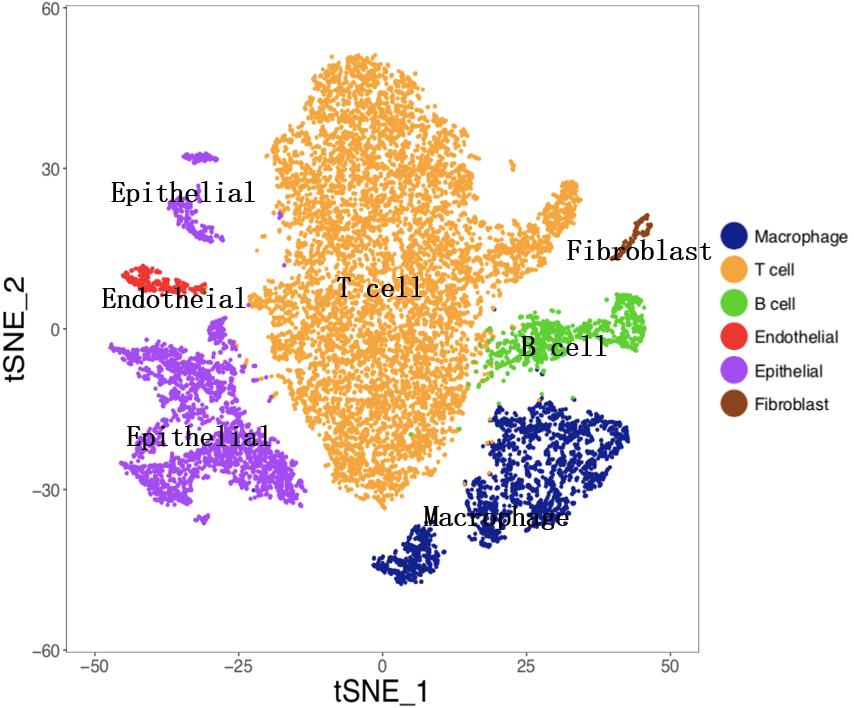
综上所述,通过标准化将数据过滤后通过PCA降维,并形成t-SNE可视化结果;降维后的矩阵进行细胞聚类分析,并计算出各类群的marker基因及表达量,以鉴定所属的细胞类型。
细胞类型鉴定后就可以进行后续深层次的拟时序分析(Pseudotime)、及SCENIC分析。后续小编将一一讲解哦~