烈冰生物单细胞测序产品另一“虎将”——ScATAC-seq一经上线就迅速走红,引起不小的轰动!很多老师也对单细胞ATAC测序产生了极大的兴趣。本期,小编就带大家一起来详细了解一下ScATAC-seq测序的原理和实验流程介绍。
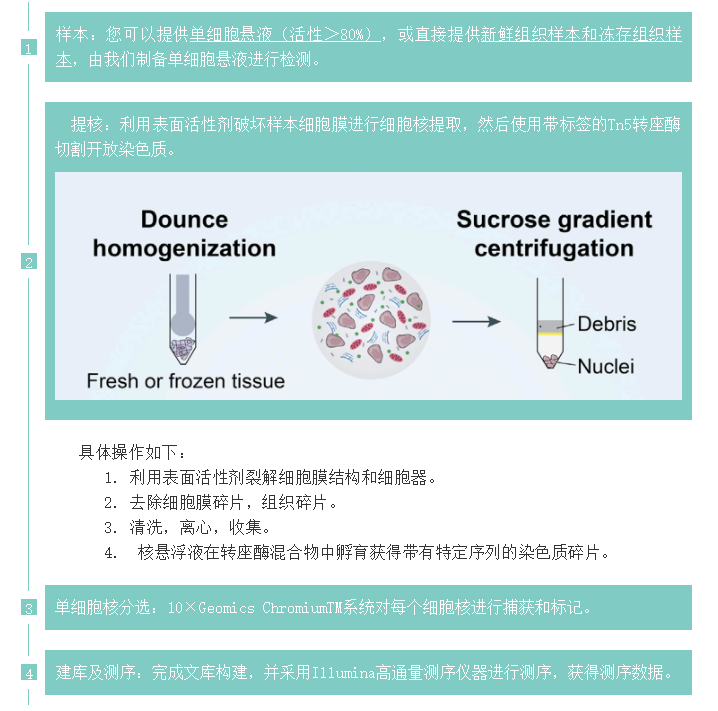
ScATAC-seq作为ATAC-seq的进阶版,能够区分实验样本的细胞类型,细胞分化过程开放性染色质有什么差异,调控了哪些信号通路等等。总的来说,ScATAC-seq能在基因组水平上帮助我们了解细胞的转录调控过程,揭示不同调控因子位点,从表观遗传学的角度来解析基因信息。ScATAC-seq在开放染色质图谱绘制,细胞分化发育,疾病的致病机制,肿瘤微环境、生物标志物等方面具有极大的应用前景。
要了解ScATAC-seq,首先我们要知道什么是ATAC-seq。ATAC-seq(Assay for Transposase-Accessible Chromatin with highthroughput sequencing)是利用转座酶研究染色质可及性的高通量测序技术,通俗来说就是基于染色质可及性的特点,将携带测序信息的Tn5转座酶插入染色质中,由于转座酶只能剪切染色质开放区域并标记DNA序列,再进行高通量测序就能获得相关基因信息。
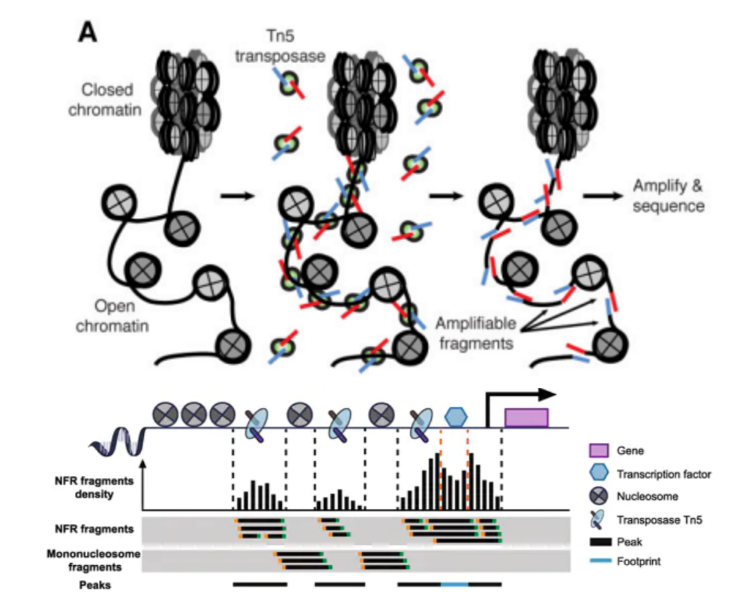

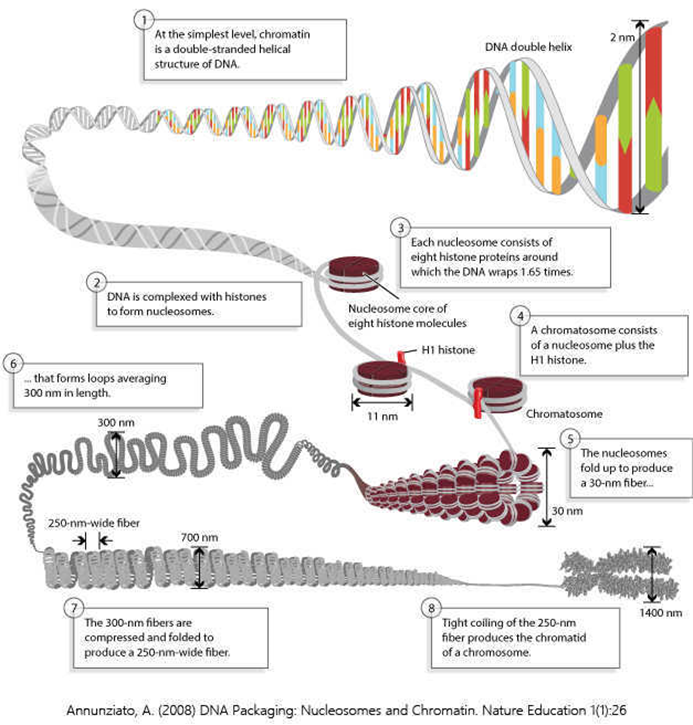
正常情况下,DNA与核小体缠绕折叠在一起形成染色质,但是DNA的复制、转录都需要将染色体的高级结构解开,然而解压并不需要打开全部染色体,只需要打开表达基因的区域,这部分打开的染色质,就叫开放染色质(open chromatin)。打开染色质,允许部分调控蛋白(比如转录因子和辅因子)与之相结合的特性,即染色质的可及性(chromatin accessibility)。
对染色质可及性探究的方法有很多,比如DNase-seq,MNase-seq,ChIP-seq,FAIRE-seq等。DNase-seq和MNase-seq方法利用限制性酶对染色质DNA进行片段化处理,该方法不能获得完整开放的染色质信息。相较于粗暴的核酸酶,ScATAC-seq利用的Tn5转座酶没有偏好性,它能够完整地将整个开放区域的序列直接捕获下来,所需的细胞数量少(最少500个起始细胞),能够在短时间内使用微量样本获得大量有效信息,且灵敏度高、实验的重复性较好。

ATAC-seq是把所有实验细胞看作了一个整体,获得所有细胞混合的基因信息。ScATAC-seq是在ATAC-seq的基础上,进行细胞核的分选和标记,通过barcode识别细胞核,解决了不同细胞群体的异质性的问题,能够检测出混杂样品测序所无法得到的异质性信息。
●●● ScATAC-seq 细胞核的捕获和标记 ●●●
上面我们介绍了转座酶是如何切割开放性染色质的。相较于ATAC-seq,ScATAC-seq在细胞核的捕获和标记方面,烈冰用10×Geomics ChromiumTM系统引入带有特定barcode标签的凝胶磁珠(beads)来标记细胞核。横向孔道中输入beads,第一列输入细胞核,细胞核和beads吸附结合后在微流控作用下向右移动,进入第二列输入的油滴中,形成油包水的结构。这时候,一个液滴=一个beads=一个细胞核=一个ATAC-seq,对酶切后的短片段进行PCR扩增反应。最后去除油滴,所有的序列混合在一起,进行上机测序。
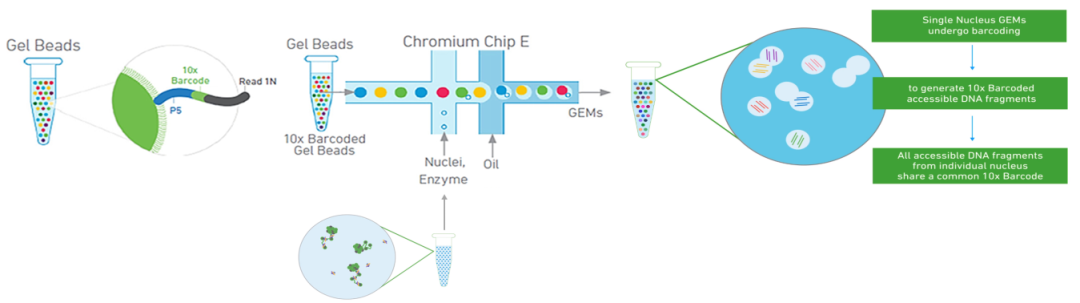
总的来说,ScATAC-Seq实验包括以下几个部分:制备单细胞悬液→细胞破碎提核→单细胞核分选→文库构建→上机测序。具体实验流程如下: