在WGBS测序中,我们选用bismark(http://www.bioinformatics.bbsrc.ac.uk/projects/bismark/)软件对fq文件进行mapping,该软件基于bowtie或者bowtie2,将BS-seq reads C→T G→A分别转化。再分别mapping到BS转化过的基因组。得到的四个alignment结果来判断最合适的unique alignment。同时软件还可以统计出甲基化的类型如CpG、CHG或者CHH等。
1、软件安装
下载地址(github):https://github.com/FelixKrueger/Bismark.git
关联软件:samtools,bowtie/bowtie2
安装方法:
git clonehttps://github.com/FelixKrueger/Bismark.git
tar-zxvf bismark_v0.15.0.tar.gz
2、软件使用方法
bismark软件分析BSSeq数据主要分为三个步骤:构建基因组并创建bowtie2索引,4次DNAmapping,统计bam文件中的信息
※构建基因组创建索引
选择bismark软件中的bismark_genome_preparation工具,需要给定bowtie2的路径以及参考基因的路径(包含fa和fai文件),操作代码如下:
bismark_genome_preparation--path_to_bowtie2/usr/local/bowtie2/--verbose/data/genomes/homo_sapiens/GRCh37/
※mapping
选择bismark工具进行mapping,需要给出基因组路径(第一步中--verbose路径),用法如下
bismark[options]{-1-2|}
例如:bismark--bowtie2--path_to_bowtie/home/novelbio/software/bowtie2/../GRCH37/-1 filtered.1.fq.gz-2 filtered.2.fq.gz-o result/
双端数据需要输入-1与-2,单端数据直接输入即可
※统计bam文件信息
选择bismark_methylation_extractor工具进行统计,用法如下:
用法:bismark_methylation_extractor[options]
测试使用代码./bismark_methylation_extractor SRR534203_filtered.fq.gz_bismark_bt2.bam-s--gzip--bedGraph--genome_folder../ath_tair10/
其中输入文件为第二部生成的bam文件,-s代表单端bam文件,-p代表双端bam文件--gzip代表对结果文件进行压缩--bedGragh代表生成带有甲基化率的bed文件
3、结果展示
※创建参考基因组
bismark将基因组的fa文件转化为两份,并分别使用bowtie2构建索引
![]()
※Mapping结果
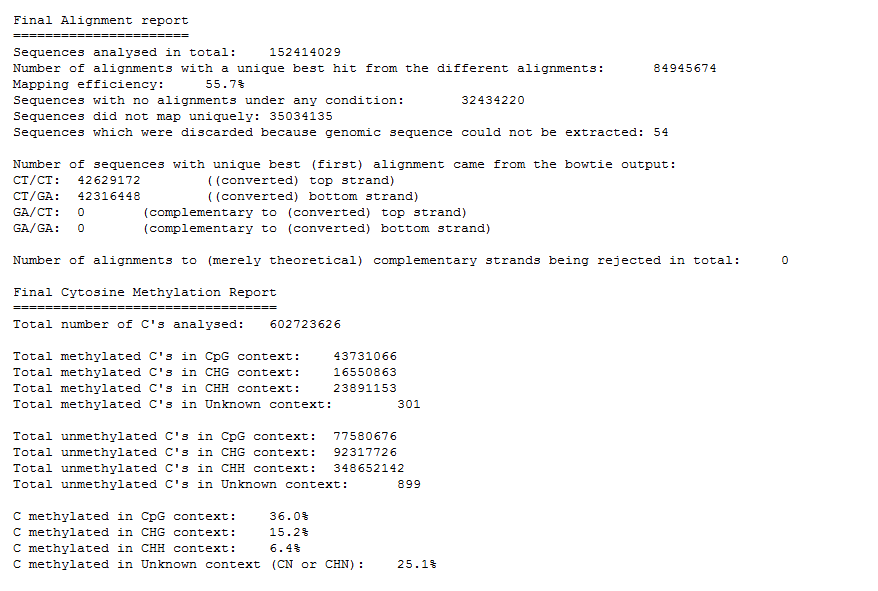
Mapping的结果中提供了reads的Mapping率,uniqueMapping情况,以及不同种类的甲基化程度
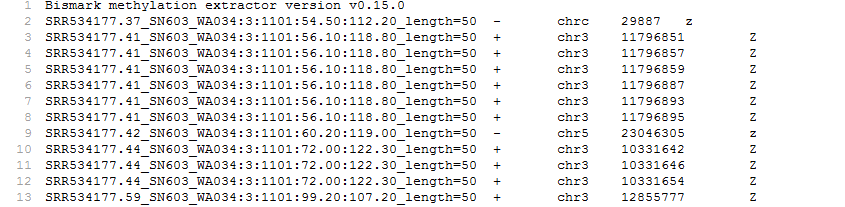
bam文件记录展示:SRR534203.2_SN608_VA028:5:1101:24.50:89.20_length=50 16 chr3 6025118 42 49M*0 0
CTCACATCAATAAAATCTAATTCAATCCTCACCTCATCTTCAAAATAAA
FGIIIHDEJHDJIIGGIGIHHCHHGCJHFGCIHFJIHHHHHHFFDDD=1
NM:i:8 MD:Z:9G1G1G0G3G0G23G0G4
XM:Z:.........x.h.hh...xh.......................hh....XR:Z:CT XG:Z:GA
在每条reads记录中提供了该位点的甲基化情况,在XM:Z:记录中,"."代表不是甲基化位点,"z/Z"代表CpG位点,其中z代表未发生甲基化位点,Z代表发生甲基化的位点,
“x/X"代表CHG位点,"h/H"代表CHH位点,“u/U"代表CN或CHN位点
在生成的mapping Report结果汇总提到的发生CpG甲基化的位点个数其实就是全部reads中出现"Z"的数量总和,其他种类甲基化的算法也是一样,甲基化率则根据(发生甲基化数量/(发生+未发生))计算
※统计结果
结果统计信息截图
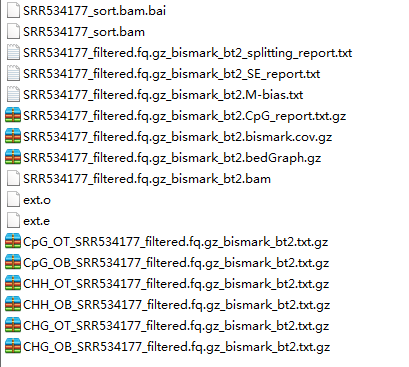
其中.bismark.cov.gz文件记录了每个甲基化位点的覆盖度,包含发生甲基化的reads数,未发生甲基化的reads数以及甲基化频率,截图如上右
bedGraph.gz文件以bed文件的格式记录了甲基化位点的甲基化频率
CpG_report.txt.gz文件记录了位点,覆盖度以及附近的位点信息
![]()
CpG_OT/OB文件记录了每一条reads的CpG甲基化情况,OT代表original top strand,OB代表original bottom strand,文件截图如下: