最近,剪接体可是着实火了一把。
这其中不得不提的就是施一公院士。
施一公何许人也,相信也不用烈小冰多做介绍。中国科学院院士、清华大学副校长、生命科学院院长、杰出的结构生物学家,更是一位有情怀有理想有态度的大师级学者。
2017年9月9日,施一公教授荣获“未来科学大奖-生命科学奖”,获奖评语中写道,该奖项旨在“表彰他在解析真核信使RNA剪接体这一关键复合物的结构,揭示活性部位及分子层面机理的重大贡献”,可谓实至名归!

10月28-29日,2017未来科学大奖颁奖典礼暨未来论坛年会在京举行,施一公教授就此次获奖发表主题演讲,表示“目前人类35%的遗传疾病或由RNA选择性剪接导致,比如视网膜色素变性、脊髓性肌肉萎缩症等”,并称“这项研究成果的意义很可能超过了我过去25年科研生涯中所有研究成果的总和”。至此, RNA可变剪接成为研究者们视线的焦点,在基础研究领域以及生物行业内引起了广泛的关注和探讨。
正如施一公院士在演讲中所说,“因为剪接过程如此之复杂,你需要七个不同的剪接体,准确的识别内含子,把外显子拼在一起,这个过程很容易出错.以至于目前为止我们认为人类已知的遗传疾病里,大约35%来自于剪接遗传”,RNA的准确剪接是一个非常复杂而困难的过程,这个过程一旦出错就很有可能会导致非常严重的遗传疾病,因此对可变剪接的研究对于相关疾病的而治疗和药物研发均有重大意义。可喜是目前已有针对剪接体的药物被美国食品药品检验局批准上市,虽然价格昂贵,但用在病人身上非常有效。对可变剪接的进一步研究有望降低医疗成本,真正造福人类社会。
(施一公院士演讲全文链接http://tech.sina.com.cn/2017-10-29/doc-ifynhhay8118096.shtml)
可变剪接是什么?小板凳排排好,烈小冰课堂开讲啦~
在高等真核生物中,基因大都以内含子-外显子交替的形式存在,当其被转录成pre-mRNA后,通过外显子的不同组合方式,可以产生一系列不同的转录本,最终使得生物体内的转录组和蛋白质组呈现多样性。在这个过程中,可变剪接起着关键的作用。在人类中,绝大多数具有蛋白编码能力的基因都能受到可变剪接的调控,最终产生多种不同功能的蛋白亚型。而异常的可变剪接也广泛涉及到了多种疾病,例如癌症等。因此,探索可变剪接事件对于进一步理解生物体内不同转录本的功能是至关重要的。
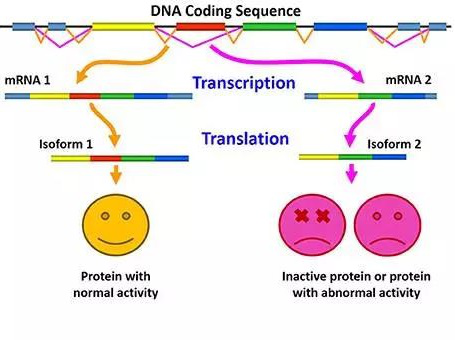
横空出世---烈冰开创高验证率可变剪接算法ASD
数据分析起家的烈冰很早便开始关注可变剪接,早在2014年即自主研发了选择性剪接分析软件(ASD,Alternative Splicing Detector,网址:http://www.novelbio.com/asd/ASD.html),协助中科院营养所冯英组对鸡细胞系的高通量测序数据进行生物信息分析,首次揭示了选择性剪接蛋白SRSF10在细胞内调节的靶底物以及其调控mRNA选择性剪接的机制,相关文章发表于权威期刊Nucleic Acids Research(IF=10.162)[1]。之后,冯老师课题组在可变剪接领域持续发力,连续发表数篇高质量文章[2-7]。

崭露头角---ASD助力多项学术研究
该算法发布后即被大量文献引用,其中不乏Nature Communications、PloS Genetics 等高分期刊[8-17]。
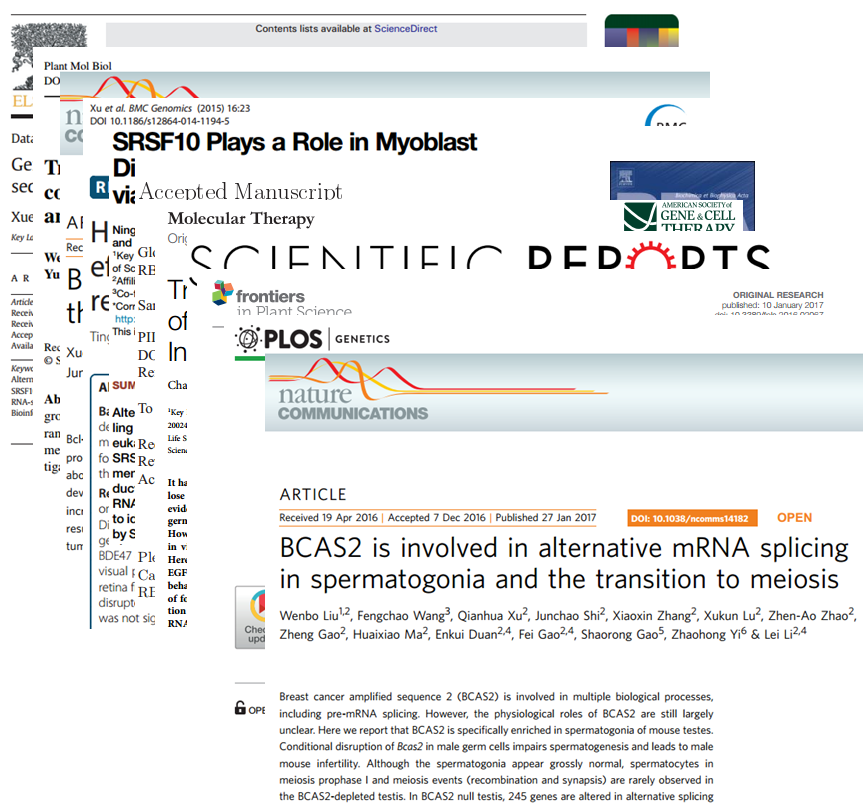
2017年1月27日,一篇题为“BCAS2 is involved in alternative mRNA splicing in spermatogonia and the transition to meiosis”的文章在线发表于Nature Communications(IF=12.124),研究团队为中国科学院动物研究所干细胞与生殖国家重点实验室李磊研究员课题组,研究结果发现BCAS2作为可变剪接调节因子,参与精原干细胞中前体RNA的正确剪接和减数分裂正常启动,ASD算法在该工作中被应用于可变剪接的检测和分析[8]。该研究首次揭示了可变剪接在精子发生过程中调控减数分裂启动这一重要事件,为哺乳动物减数分裂启动研究提供了新的思路,并为可变剪接生理功能研究提供了重要理论依据。

仅用于动物研究?NO!植物领域依旧所向披靡!
3月8日,PLoS Genetics杂志在线发表了中国科学院上海生命科学研究院植物生理生态研究所方玉达研究组题为“Depletion of Arabidopsis SC35 and SC35-like serine/arginine-rich proteins affects the transcription and splicing of a subset of genes”的研究论文,文章借助ASD算法对差异可变剪接进行检测和分析,研究结果揭示拟南芥核小斑中SC35及其类似蛋白(SCL蛋白)可调控部分基因的转录和剪接,从而影响植物生长发育的分子生物学机制[9]。
5月份,来自西北农林科技大学的李海峰老师团队和烈冰生信团队合作,以模式植物二穗短柄草为材料,采用目前速度最快的NGS测序平台Ion Proton,进行深度转录组测序,深入系统全面进行高温胁迫相关差异基因、差异转录因子、差异可变剪接等项目的分析[10]。其中,ASD再次为植物领域RNA-seq数据深入分析提供强有力的支持。该文章的发表也用事实证明了ASD算法的多平台兼容性,不管是Illumina平台,还是Ion Proton平台,ASD都能对其产生的测序数据进行高校准确的分析,助力不同平台的数据挖掘。
再度升级---验证&检出率最高的CASH惊艳现世
虽然ASD算法已获得了众多学者的信任和支持,但是,烈冰怎么可能止步不 前!
2017年4月6号,烈冰研发总监宗杰博士率生物信息研发团队开发的“进阶级”可变剪接分析算法CASH(Comprehensive AS Hunting,网址:https://sourceforge.net/projects/cash-program/)以“CASH: a constructing comprehensive splice site method for detecting alternative splicing events”为题在线发表于生物信息类期刊Briefings in Bioinformatics(IF=5.134)。通过与Cuffdiff,MISO,DEXSeq和rMATS等已有算法进行比较后发现,无论在有生物学重复还是无生物学重复样本中,CASH都显著提升了样本之间差异可变剪接事件的检测能力,尤其是新的可变剪接事件,验证准确率高达70%!在针对不同测序深度数据的测试中,CASH始终表现出优于其他算法的检测率。即使是在低数据量下,CASH依旧力压其他算法,始终维持着极高的敏感性及特异性。
这是继ASD算法后,烈冰生物发表的第二篇可变剪接检测算法类文章,创下国内同类算法的又一里程碑,在创新型企业自主研发算法攻坚之路上再下一城!


CASH算法发表之后也引起了研究界的广泛关注。中科院上海植物逆境中心主任朱健康院士于7月份发表在Nature Communications上的研究以模式植物拟南芥为研究材料,发现了一个在植物对外源脱落酸(ABA)胁迫的应答中起关键作用的RNA剪接因子ROA1/RBM25, CASH算法在其中发挥了重要的作用[18]。

朱健康院士的研究再次证明了对植物进行可变剪接研究的重要性,也为CASH算法在植物可变剪接中的应用提供了强有力的支持。
看到我们自主研发的两个算法得到这么多科研前辈的认可,烈小冰受到巨大的鼓励,并感到深深的自豪。我们一次又一次地用行动证明,研发实力就是烈冰的核心竞争力,烈冰一直在踏踏实实地为老师解决实际问题,并不断超越自我,为科研的发展贡献自己的一份力量。
此次烈冰自主研发的双可变剪接算法ASD和CASH不仅在医学领域,还在农学领域为广大科研工作者的可变剪接研究提供了高效准确的分析工具,实现了样本间差异可变剪接的高检出率和验证率,在可变剪接算法史上写下辉煌的一笔。
烈冰在自己最擅长的领域匠心筑梦,凭借实力助力您的科学研究。想了解更多信息,请联系您的区域业务经理进行咨询哦~
1. Zhou X, et al. Nucleic Acids Res. 2014.
2. Zhou X, et al. Genomics Data. 2014.
3. Zhou X, et al. Nat Commun. 2014.
4. Wei N, et al. Cell Reports. 2015.
5. Cheng Y, et al. Mol Cell Biol. 2016.
6. Luo C, et al. Can Res. 2017.
7. Liu L, et al. Oncogene. 2017.
8. Liu W, et al. Nat Commun. 2017.
9. Yan Q, et al. PloS Genet. 2017.
10. Chen S, et al. Front Plant Sci. 2017.
11. Cheng T, et al. Sci Rep. 2017.
12. Xu W, et al. Plant Mol Biol. 2014.
13. Xu T, et al. BMC Genomics. 2015.
14. Mai S, et al. BBA - Gene Regul Mech. 2016.
15. Hu B, et al. Sci Rep. 2016.
16. Miao X, et al. Gene. 2016.
17. Wu C, et al. Mol Ther. 2017.
18. Zhan X, et al. Nat Commun. 2017.